Molekularbiologische Services und Biofilm-Forschung
Unser Leistungsschwerpunkt ist die Analyse medizinischer Biofilme durch Fluoreszenz in situ Hybridisierung (FISH) und Nukleinsäure-Amplifikationsverfahren (PCR, Sequenzierung, Mikrobiom-Analysen). Durch die Kombination von Visualisierung und Genom-/Mikrobiom-Analysen erhalten wir völlig neue Einblicke in Infektionslandschaften und Biofilme. Für unser diagnostisches Leistungsspektrum sehen Sie bitte: www.moter-diagnostics.com/diagnostische-leistungen
Das Spektrum unserer Dienstleistungen umfasst den Nachweis von Mikroorganismen aus in vitro Experimenten und in Gewebeproben. Ein Schwerpunkt liegt auf der Quantifizierung und Analyse von Biofilmen. Dabei zeigt die FISH die Effektivität von anti-Biofilm-Substanzen oder beschichteten Oberflächen medizinischer Produkte (z. B. Implantate) gegen Mikroorganismen.
Visualisierung und Quantifizierung von Biofilmen
Messung von mikrobiellen Biofilmen
Fluoreszenz in situ Hybridisierung (FISH) stellt Biofilme mikroskopisch dar, wodurch sie in Ausdehnung, Dicke und Lokalisation messbar werden.
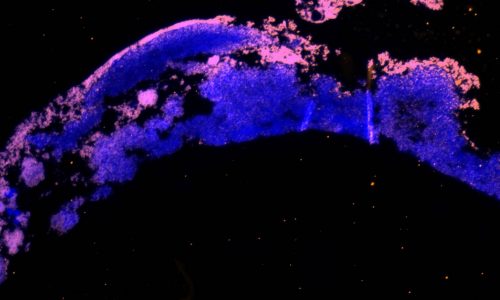
FISH eines bakteriellen Biofilms (in blau der Nukleinsäurefarbstoff DAPI, der alle Bakterien zeigt, in magenta die FISH-positiven Bakterien).
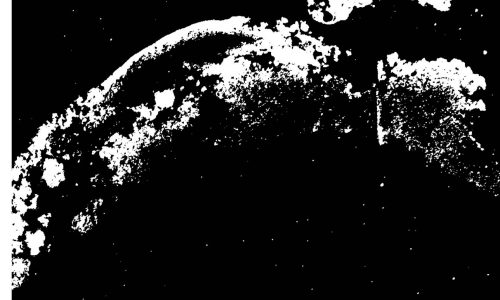
Segmentierung des Bildes durch digitale Bildanalyse zur Quantifizierung des FISH-positiven Anteils im Biofilm.
Standardisierte FISH-Testsysteme
für antimikrobielle Substanzen
Fluoreszenz in situ Hybridisierung zur Messung der Effektivität von antimikrobiellen Substanzen
Fluoreszenz in situ Hybridisierung (FISH) ist ein molekularbiologisches Verfahren, das in der Lage ist, die Wirksamkeit von antimikrobiellen Substanzen auf Biofilme zu visualisieren und dadurch direkt messbar zu machen.
Damit eignet sich die FISH auch für die Testung von Anti-Infektiva. So wird Kultur-unabhängig der Effekt mit räumlicher Auflösung quantifizierbar: Wo wirkt die Substanz? Wie wirkt sie? Verbleiben überlebende Mikroorganismen? Wie Zeit- und Dosis-abhängig ist der Abtötungsprozess im Biofilm?
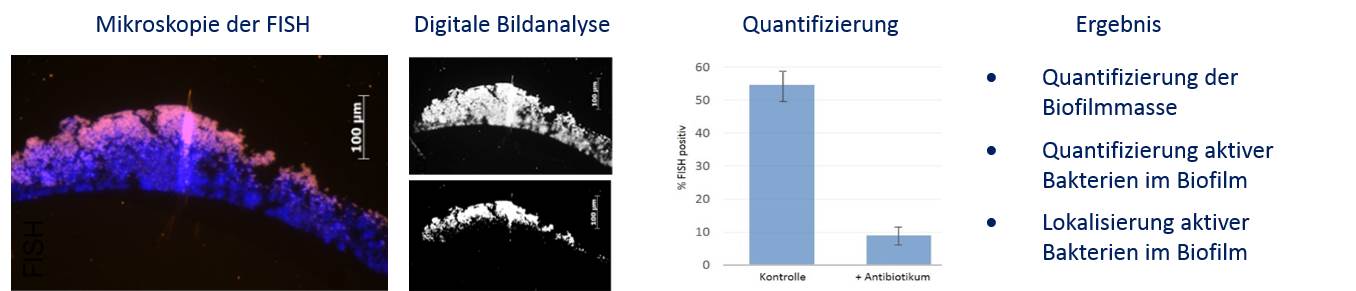
FISH eines Staphylokokken-Biofilms. Nachweis der in situ Wirksamkeit von Antibiotika auf Biofilme (DAPI, blau). Im Vergleich zur Kontrolle nimmt der Anteil an aktiven, FISH-positiven Bakterien (Magenta) nach Behandlung mit Antibiotika ab.
Testung der antimikrobiellen Eigenschaften
medizinischer Oberflächen
Visualisierung und Quantifizierung von Biofilmen durch FISH
Fluoreszenz in situ Hybridisierung (FISH) visualisiert und misst den Effekt von antimikrobiell beschichteten Oberflächen gegen Mikroorganismen und Biofilme. So wird Kultur-unabhängig der Effekt z.B. von beschichteten Implantatoberflächen gegen Bakterien mit räumlicher Auflösung quantifizierbar.

In Methacrylat eingebettete Katheterquerschnitte mit anhaftendem bakteriellen Biofilm.
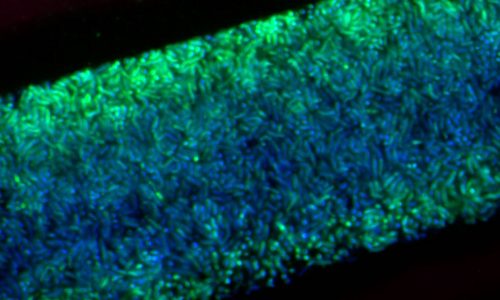
Mikrobieller Biofilm auf einer Oberfläche mit Schichten unterschiedlich aktiver Bakterien. Aktive Bakterien zeigen ein starkes FISH-Signal (grün), während inaktive Bakterien nur durch den Nukleinsäurefarbstoff DAPI (blau) angefärbt werden.
FISH-Analyse von Mikroorganismen - Custom FISH
Neuentwicklung individueller FISH-Sonden und -Testsysteme
Für Forschungsanwendungen entwickeln und optimieren wir nach Bedarf individuelle FISH-Sonden und -Testsysteme. Dies umfasst Flow-Chamber-Systeme (Durchflusskammern) bis hin zu Gewebeproben.
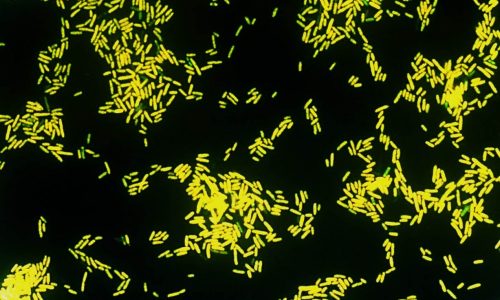
FISH von Escherichia coli (Gram-negatives Stäbchen).
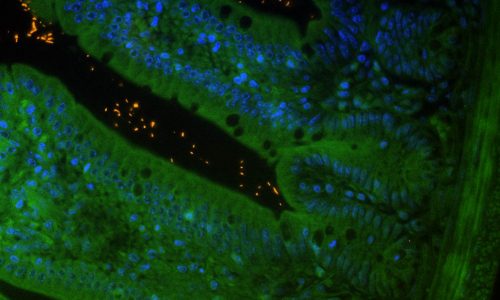
FISH-Analyse von Darm-Mikroorganismen im Lumen eines murinen Darm-Querschnitts. Gewebshintergrund in grün (Autofluoreszenz), Nukleinsäuren in blau durch den Nukleinsäurefarbstoff DAPI, FISH-positive Bakterien in orange (EUB338-Sonde).
Molekulare Identifikation von Mikroorganismen
16S rRNA Gen PCR-Amplifikation und Sanger Sequenzierung
Für die Identifikation von Reinkulturen oder Mikroorganismen in Geweben bieten wir die klassische pan-bakterielle Amplifikation des 16S rRNA Gens mit nachfolgender Sequenzierung nach Sanger an. Bei der PCR (Polymerase Chain Reaction) werden Teilbereiche der Erbsubstanz der Mikroorganismen (z.B. Teile des 16S rRNA-Gens) vervielfältigt und anschließend durch Sequenzierung die Identität der Mikroorganismen analysiert.
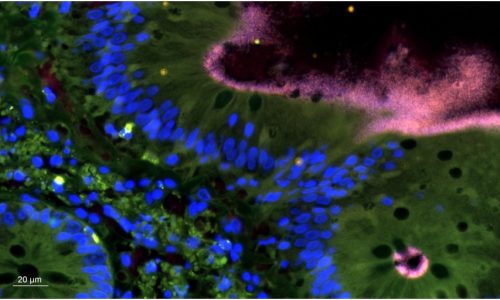
Gewebsprobe mit Besiedlung durch Brachyspira sp. nachgewiesen durch FISH.
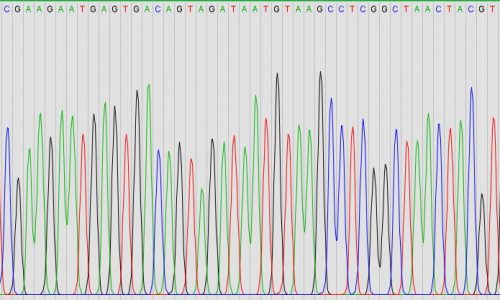
Identifikation von Brachyspira aalborgi aus Schnitten der Gewebsprobe durch PCR-Amplifikation und Sanger Sequenzierung.
MG-FISH
Microbiome-guided (MG) FISH
Bei gemischten Biofilmen kombinieren wir die FISH mit Mikrobiomanalysen, die alle in einer Probe vorhandenen Mikroorganismen (das gesamte Mikrobiom) analysieren.
Mikrobielle DNA lässt sich sehr sensitiv und Kontaminations-arm aus in Methacrylat-eingebetteten Proben amplifizieren. Die Interpretation der FISH gemeinsam mit den Sequenzergebnissen erhöht die Treffsicherheit bedeutend.
So erhalten die Mikrobiom-Daten eine räumliche Auflösung im Probenzusammenhang:
- Mengenverteilung der beteiligten Mikroorganismen
- Lokalisation am und im Gewebe
- Identifikation von Schlüsselpathogenen
- räumlicher Zusammenhang in der Probe (erste Besiedler von Implantaten? Gewebe-invasive Pathogene?)
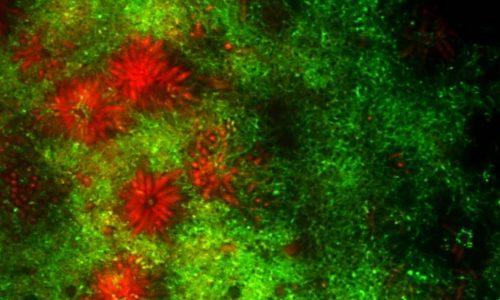
FISH eines oralen Multispezies-Biofilms.
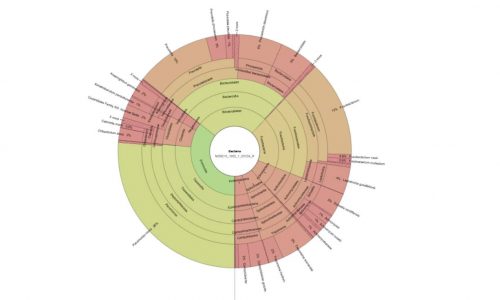
Mikrobiomanalyse aus Schnitten des oralen Biofilms.
